Boletín UNAM-DGCS-553
Cuernavaca, Morelos
Pie de foto al final del
boletín
ESCUDRIÑAN CON
GENÉTICA Y BIOINFORMÁTICA
·
Se han aislado bacterias que causan
enfermedades, pero se desconoce su contexto ecológico, afirmó Pablo Vinuesa,
del Centro de Ciencias Genómicas de
·
En el campus Morelos, el microbiólogo y su
grupo han caracterizado microorganismos en ríos y suelos conservados y contaminados
cercanos a Cuernavaca
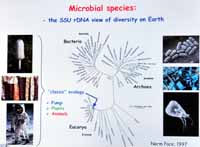
La diversidad de bacterias es un universo aún por descubrir, pues se
ignoran casi todas las características de su entorno ecológico y sus relaciones
evolutivas, afirmó el microbiólogo Pablo Vinuesa, investigador del Centro de
Ciencias Genómicas (CCG) de
“Las bacterias que tradicionalmente han interesado a la ciencia son
pocas en relación con su gran diversidad, se relacionan con enfermedades y
tienen una gran importancia clínica, pero se han estudiado aisladas, de forma
sesgada, con desconocimiento al aspecto
ecológico”, señaló.
Para escudriñar el ambiente natural de las bacterias en ecosistemas
conservados y contaminados cercanos a Cuernavaca, Vinuesa desarrolla un
proyecto de investigación para estudiar dos grupos de bacterias --rizobios y
micobacterias--, en ecosistemas naturales en buen estado de conservación, así
como en hábitats contaminados por el ser humano que modifican sus poblaciones asociadas
con aguas negras y suelos dañados.
“La presencia de los seres humanos no solamente ha afectado a otros
animales y a las plantas, sino también a microorganismos fundamentales como las
bacterias, de las que sabemos muy poco pero fundamentales para la vida en el
planeta”, señaló Vinuesa, especialista del CCG en microbiología ambiental y
evolutiva.
El investigador añadió que plantas y animales son una parte ínfima de
la diversidad, y son los microbios la base del funcionamiento del planeta.
“Son los que inventaron la fijación del nitrógeno, la fotosíntesis y el
sistema de transformación de la energía del Sol para aprovechar el agua y, a
partir de ahí, sintetizar azúcares.
Nosotros, por ser organismos macroscópicos tenemos un cierto rasgo de
arrogancia, pero explorando el universo de las bacterias podemos responder a
preguntas como por qué hay tantas enfermedades o por qué un suelo de cultivo ha
dejado de ser fértil”, advirtió.
Marcadores moleculares
Para su estudio, Vinuesa y sus colaboradores muestrean bacterias y las
llevan al laboratorio para estudiarlas a nivel molecular y genómico.
“Desarrollamos una aproximación a escala genómica para tratar de
identificar marcadores moleculares, fragmentos de genes que contienen
información muy valiosa para este tipo de estudios de diversidad, tanto a nivel
de genética de poblaciones como de filogenias, para conocer nuevos linajes de
bacterias que va recuperando el ambiente, pues queremos saber de qué microorganismos
se trata”, señaló el académico.
La forma más eficiente de realizar ese estudio implica un análisis
comparativo de secuencias de distintos genes.
“Tradicionalmente, se ha seleccionado un solo gene, el 16 S ribosomal,
porque es el que tenemos todos los organismos conocidos y es fundamental para
los ribosomas, que son las máquinas que hacen la síntesis de proteínas. Pero es
mucho más poderosa la alternativa de utilizar múltiples genes, sobre todo si
codifican para proteínas, y utilizarlos con métodos analíticos avanzados”,
consideró.
Para avanzar en la caracterización de bacterias en ecosistemas tanto
conservados como contaminados, Vinuesa desarrolló un marco conceptual y
computacional.
“Se trata de una tubería de análisis que utiliza la información de los
genomas para identificar segmentos de genes que sean particularmente
informativos para develar las relaciones evolutivas entre ese grupo de
organismos que queremos estudiar, aclaró.
Estos análisis, que requieren de bioinformática para almacenar y
analizar gran cantidad de datos, implican tomar muestras, analizar su ADN y
elegir marcadores moleculares que revelen características de las bacterias en
consideración de su entorno.
“Hay un proceso de obtención de materiales representativos con una
aproximación computacional”, señaló.
Rizobios y micobacterias
Las bacterias rizobios están asociadas simbióticamente con leguminosas,
pero no dependen de esas plantas para su sobrevivencia.
En tanto, las micobacterias se relacionan con enfermedades como la
tuberculosis y la lepra, pero las causantes de esas enfermedades son sólo dos
variantes de una gran variedad de microorganismos, señaló el investigador.
“Las rizobios se han estudiado básicamente por su asociación con
leguminosas como el frijol, soya, alfalfa y garbanzo, pero son simbiontes
obligados, pueden vivir en el suelo, en un plásmido (que es un anillo de ADN) o
desarrollar una transferencia genética horizontal”, dijo Vinuesa.
“La diversidad bacteriana es inmensa, y con esta investigación podemos
adentrarnos también en la ecología de bacterias en el suelo”, aclaró.
Tras extraer el ADN de bacterias, Vinuesa y su equipo generan librerías
de fragmentos de las bacterias, útiles para conocer la diversidad mediante estudios
analíticos y comparativos, necesarios para recrear el contexto ecológico de las
bacterias.
--oOo—
Foto 01.
La diversidad de bacterias es un universo aún por descubrir,
afirmó el microbiólogo Pablo Vinuesa, investigador del Centro de Ciencias
Genómicas (CCG) de la UNAM.
Foto 02
Para estudiar el ambiente natural de las bacterias en
ecosistemas conservados y contaminados, Vinuesa desarrolla un proyecto con dos
grupos de bacterias --rizobios y micobacterias--.